Translated by 
机器之心 / 2019.07.08 / Original link
2019年7月,国际期刊Cancer Medicine以封面文章的形式,刊登了点内科技、复旦大学附属华东医院、上海交通大学等多单位的最新联合研究成果,《基于3D深度学习自动预测肺腺癌EGFR突变状态》,研究显示预测精准度超过传统影像组学。
基于3D深度学习自动预测肺腺癌EGFR突变状态,预测精准度超过传统影像组学

肺癌是肿瘤相关死亡的首因[1],肺癌患者中80%为非小细胞肺癌(NSCLC),腺癌为其最常见的病理类型[2]。近数十年来基于基因组学而发展的针对驱动基因的靶向治疗,如靶向特定表皮生长因子受体(EGFR)突变的酪氨酸激酶抑制剂(TKI)吉非替尼、靶向ALK基因的TKI 克唑替尼等,已成为肺癌精准医学不可或缺的重要组分[3]。然而,对于无EGFR突变或非ALK阳性的肺癌患者,靶向治疗却并无临床获益,有时还可能导致PFS缩短以及不必要的医疗支出[4],因此,选择靶向药物前需要提前明确驱动基因状态。此外,EGFR TKI治疗期间可能出现EGFR T790突变而导致耐药,出现病情进展,因此,也需要动态监测相关基因的突变状态以调整治疗药物[5]。
对活检或外科手术切除后的样本进行突变检测是明确EGFR突变类型的标准手段,但取样的侵入性、为监测治疗所必须的反复取样等问题,以及DNA质量不佳、肿瘤异质性、患者床位周转时间、检测费用等诸多难题限制了分子检测的推广,也一定程度上影响了肺癌精准医疗在临床实践中的推广规模[6-9]。
肿瘤的表型源于特定的基因型,因此,通过甄别特定表型来预测基因型是一种潜在可行的手段。已有研究显示,特定的影像组学特征与EGFR突变类型相关[10,11]。然而,传统影像组学面临诸多挑战,如在检测、分隔、提取特征等过程中大量依赖手工操作,费时费力;不同读片者之间的可复制性较低。
近年来以深度卷积神经网络为代表的深度学习在医学图像计算方面显示出令人难以置信的优越表现,并在很大程度上降低了人工操作的必要性。此前,点内科技联合复旦大学附属华东医院和上海交通大学SJTU-UCLA机器感知与推理联合研究中心等利用3D深度学习技术,成功证明了3D深度学习在预测肺腺癌侵袭性方面的有效性和效能,准确率高达88%[12]。受其鼓舞,点内科技再次与华东医院李铭、Hua Yanqing团队、以及上海交通大学SJTU-UCLA机器感知与推理联合研究中心、上海第十人民医院、同济大学同济医院等合作,探索了3D深度学习在基于CT影像预测EGFR突变方面的潜能。研究结果近期发表于 Cancer Medicine (IF=3.2,Toward automatic prediction of EGFR mutation status in pulmonary adenocarcinoma with 3Ddeep learning),华东医院李铭、滑炎卿教授为论文的共同通讯作者,华东医院赵伟和上海交大杨健程为共同第一作者。
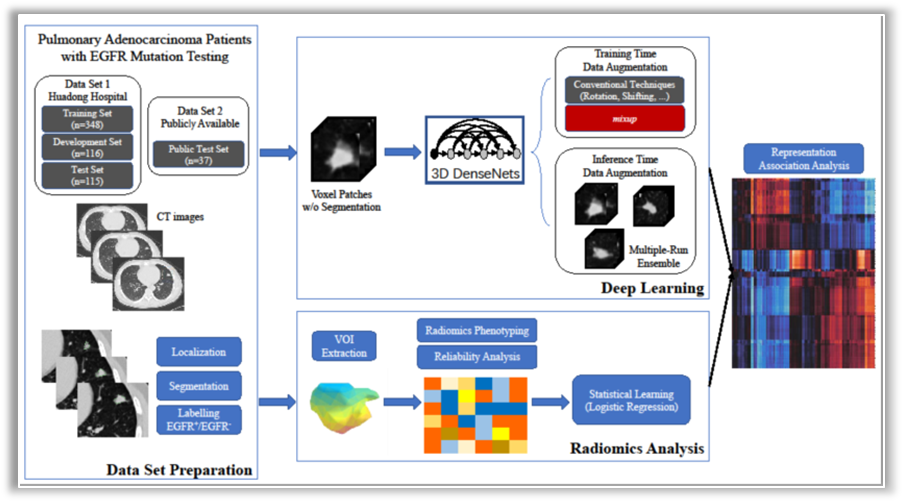
研究的影像数据来自华东医院CT数据库(HdH数据库579例,包括训练集348例、开发集116例和测试集115例)。为测试所建模型的通用性,从独立的肿瘤公共影像库TCIA选取37例肺结节也作为测试集。全部病例均由人工进行定位、分割和标记EGFR突变/野生型状态。通过训练集采用3D DenseNets深度学习方法训练,采用强大的数据扩增技术mixup以提升规范化/正则化(regularization),通过有监督的端到端学习模型完成拟合。与此同时,对全部影像数据进行传统的影像组学分析,与3D深度学习对照,计算成对关联系数分析二者的关联性。
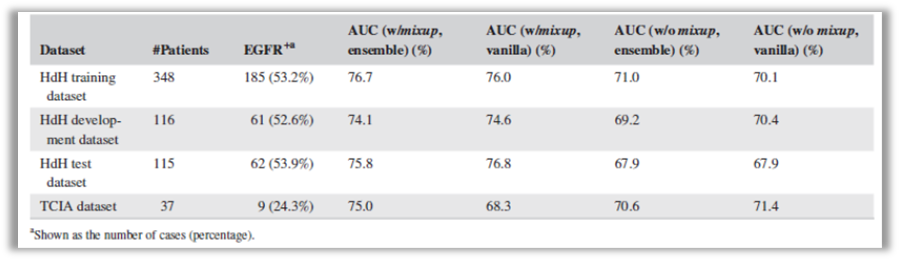
结果显示,3D深度学习在预测EGFR突变状态方面明显优于传统的影像组学方法(P=0.021),3D深度学习对HdH数据库测试集和公共测试集测试的AUC分别为75.8%和75.0%(表1)。更重要的是,不同于传统影像组学要求人工对感兴趣的病灶部位进行分割,非常耗时耗力,3D深度学习的兴趣部位与肺结节病变部位有良好的一致性。
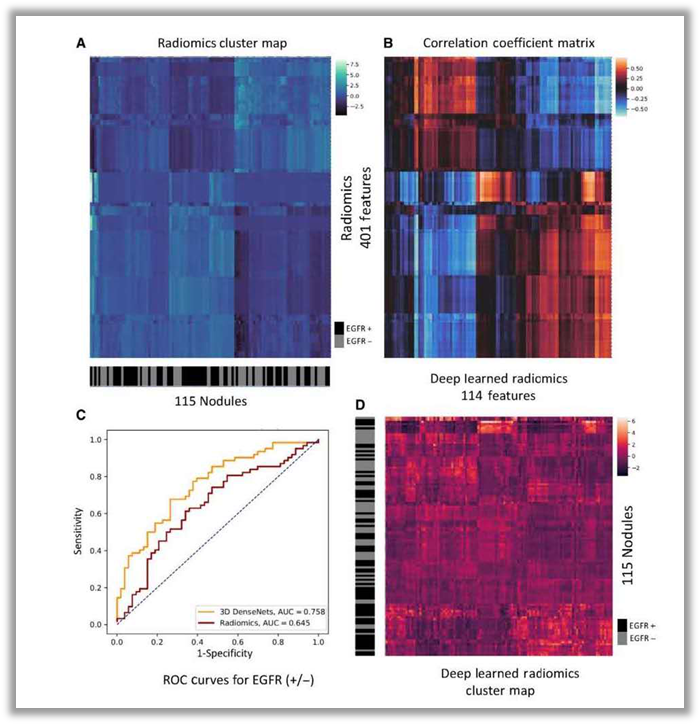
研究者对提取的401个传统影像组学特征(上图A)与从3D DenseNets提取的114个深度学习影像组学特征(上图C)进行分析发现,3D深度学习方法提取的特征较通过人工提取的特征更具代表性,因此聚类分析的结果更佳。研究首次用矩阵的方法进一步比较了深度学习特征与传统影像组学特征(上图B),分析发现,深度学习特征与传统影像学之间存在着强关联,从分类性能的ROC曲线方面,深度学习特征的敏感性和特异性更高,这说明深度学习通过增强的影像组学方式,从鲁棒性(robustness)、紧密度(compactness)和表达能力(expressiveness)方面可以发挥强大的预测效能。
研究者发展的这一深度学习体系实现了非侵入性、自动预测肺腺癌EGFR突变型,从而通过明确EGFR靶向治疗的获益人群,辅助肺腺癌的临床治疗决策。研发过程中整合了近期关于深度监督学习的一些进展,比如深度连接、mixup技术等,从而显著降低了过度拟合等风险。由于这一方法因无需对肺结节进行精细的分隔,因此非常节省劳动力,同时,因为采用的学习算法的增强特性,可望获得更加稳定的表现。
研究者称,未来还需进一步对成果进行验证,比如本研究EGFR突变是通过ARMS-PCR检测的,未来还需对有二代测序的EGFR突变数据的样本进行验证;目前的模型仅纳入了CT影像信息,未来还需整合更多的临床信息如病理、血检验结果、蛋白组学等。

(上图为2019年7月刊Cancer Medicine封面)
【参考文献】
-
Bray F, Ferlay J, Soerjomataram I, Siegel RL, Torre LA, Jemal A. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 2018;68(6):394-424.
-
Ettinger DS, Wood DE, Aisner DL, et al. Cell lung cancer, Version 5.2017, NCCN Clinical Practice Guidelines in Oncology. J Natl Compr Canc Netw. 2017;15(4):504-535.
-
NCCN临床实践指南:非小细胞肺癌(2019.V4)
-
Mok TS, Wu Y-L, Thongprasert S, et al. Gefitinib or carboplatin-paclitaxel in pulmonary adenocarcinoma. N Engl J Med.2009;361(10):947-957.
-
中国临床肿瘤学会肿瘤生物标志物专家委员会《中国非小细胞肺癌患者EGFR T790M基因突变检测专家共识》制定专家组 中国非小细胞肺癌患者EGFR T790M基因突变检测专家共识 中华医学杂志2018,98(32:2544-51)
-
Bedard PL, Hansen AR, Ratain MJ, Siu LL. Tumour heterogeneity in the clinic. Nature. 2013;501(7467):355-364.
-
Lindeman NI, Cagle PT, Beasley MB, et al. Molecular testing guideline for selection of lung cancer patients for EGFR and ALK tyrosine kinase inhibitors: guideline from the College of American Pathologists, International Association for the Study of Lung Cancer, and Association for Molecular Pathology. J Mol Diagn. 2013;15(4):415-453.
-
Kim TO, Oh IJ, Kho BG, et al. Feasibility of re-biopsy and EGFR mutation analysis in patients with non-small cell lung cancer. Thorac Cancer. 2018;9(7):856-864.
-
Kobayashi K, Naoki K, Manabe T, et al. Comparison of detection methods of EGFR T790M mutations using plasma, serum, and tumor tissue in EGFR-TKI-resistant non-small cell lung cancer. Onco Targets Ther. 2018;11:3335-3343.
-
Yip S, Kim J, Coroller TP, et al. Associations between somatic mutations and metabolic imaging phenotypes in non‐small cell lung cancer. J Nucl Med. 2017;58(4):569-576.
-
Rios Velazquez E, Parmar C, Liu Y, et al. Somatic mutations drive distinct imaging phenotypes in lung cancer. Can Res. 2017;77(14):3922-3930.
-
Zhao W, Yang J, Sun Y, et al.3D Deep Learning from CT Scans Predicts Tumor Invasiveness of Subcentimeter Pulmonary Adenocarcinomas. Cancer Res. 2018 Dec 15;78(24):6881-6889.